Erforderliche Pakete laden
library(rms) # binär logistische Regression
library(MASS) # Modellvereinfachung
library(DAAG) # Kreuzvalidierung
library(pROC) # ROC-Analyse
Datensatz einlesen und Modell spezifizieren
# Datensatz 'data': pro Versuchsperson eine Zeile
data <- read.table("https://mmi.psycho.unibas.ch/r-toolbox/data/LR1.txt", header=TRUE)
# Modell spezifizieren
model <- Symptom~Alter+Geschlecht
Binär logistische Regression
library(rms)
variablen <- all.vars(model)
data2 <- na.omit(data[, variablen, drop=FALSE])
data2[,variablen[1]] <- as.factor(data2[,variablen[1]])
m <- lrm(model, data=data2, x=TRUE, y=TRUE)
# Wichtigkeit der Prädiktoren/Faktoren
Prädiktoren <- anova(m)
# Pseudo R2
model0 <- as.formula(paste(variablen[1], "~1", sep="")) # Nullmodell
m0<-glm(model0, data = data2, family=binomial(link=logit))
n <- nrow(na.omit(data2[,variablen]))
Cox.and.Snell <- as.numeric(1-exp((2/n)*(logLik(m0)-logLik(m))))
Negelkerke <- as.numeric((1-exp((2/n)*(logLik(m0)-logLik(m))))/(1-exp(logLik(m0)*(2/n))))
McFadden <- as.numeric(1-(logLik(m)/logLik(m0)))
R2 <- data.frame(Cox.and.Snell,Negelkerke,McFadden)
# Klassifikation
pr <- predict(m, type="fitted"); pr.nominal <- pr
pr.nominal[pr < .5] <- levels(data2[,variablen[1]])[1]
pr.nominal[pr >= .5] <- levels(data2[,variablen[1]])[2]
pr.nominal <- factor(pr.nominal, levels= levels(data2[,variablen[1]]))
klass.tab <- xtabs(~ data2[,variablen[1]] + pr.nominal)
# Nach Zufall zu erwartender Anteil richtiger Klassifikationen
Zufall <- as.numeric(rowSums(klass.tab) %*% rowSums(klass.tab)/sum( (klass.tab))^2)
# Anteil richtig klassifiziert
ant.ri.kl <- sum(diag(klass.tab))/sum(klass.tab) # insgesamt
Prozent.korrekt <- diag(klass.tab)/apply(klass.tab,1,sum)*100 # pro Zeile
klass.tab <-cbind(klass.tab, "korrekt in %"=Prozent.korrekt)
names(dimnames(klass.tab)) <- c("beobachtet", "vorhergesagt")
# Output
list("Tests für das Gesamtmodell und pro Parameter"=m, "Odds Ratios: exp(B)"=exp(m$coeff), "Tests pro Prädiktor/Faktor"=Prädiktoren, "Pseudo R2"=R2, Klassifikation=klass.tab, "Anteil richtig klassifizierter Fälle"=ant.ri.kl, "Nach Zufall zu erwartender Anteil richtig klassifizierter Fälle" = Zufall)## $`Tests für das Gesamtmodell und pro Parameter`
## Logistic Regression Model
##
## lrm(formula = model, data = data2, x = TRUE, y = TRUE)
##
## Model Likelihood Discrimination Rank Discrim.
## Ratio Test Indexes Indexes
## Obs 40 LR chi2 16.54 R2 0.451 C 0.849
## negativ 20 d.f. 2 g 2.104 Dxy 0.698
## positiv 20 Pr(> chi2) 0.0003 gr 8.199 gamma 0.703
## max |deriv| 7e-08 gp 0.350 tau-a 0.358
## Brier 0.162
##
## Coef S.E. Wald Z Pr(>|Z|)
## Intercept -6.3531 2.7793 -2.29 0.0223
## Alter 0.1581 0.0616 2.56 0.0103
## Geschlecht=weiblich -3.4898 1.1992 -2.91 0.0036
##
##
## $`Odds Ratios: exp(B)`
## Intercept Alter Geschlecht=weiblich
## 0.001741315 1.171234491 0.030506119
##
## $`Tests pro Prädiktor/Faktor`
## Wald Statistics Response: Symptom
##
## Factor Chi-Square d.f. P
## Alter 6.58 1 0.0103
## Geschlecht 8.47 1 0.0036
## TOTAL 8.79 2 0.0123
##
## $`Pseudo R2`
## Cox.and.Snell Negelkerke McFadden
## 1 0.3385871 0.4514495 0.2981885
##
## $Klassifikation
## vorhergesagt
## beobachtet negativ positiv korrekt in %
## negativ 15 5 75
## positiv 4 16 80
##
## $`Anteil richtig klassifizierter Fälle`
## [1] 0.775
##
## $`Nach Zufall zu erwartender Anteil richtig klassifizierter Fälle`
## [1] 0.5
Modellvereinfachung
library(MASS)
m.glm <- glm(model, data=data2, family='binomial')
stepAIC(m.glm, direction="both")## Start: AIC=44.92
## Symptom ~ Alter + Geschlecht
##
## Df Deviance AIC
## <none> 38.917 44.917
## - Alter 1 48.869 52.869
## - Geschlecht 1 53.023 57.023##
## Call: glm(formula = Symptom ~ Alter + Geschlecht, family = "binomial",
## data = data2)
##
## Coefficients:
## (Intercept) Alter Geschlechtweiblich
## -6.3531 0.1581 -3.4898
##
## Degrees of Freedom: 39 Total (i.e. Null); 37 Residual
## Null Deviance: 55.45
## Residual Deviance: 38.92 AIC: 44.92
k-fold Cross Validation
k <- 10 # k eingeben
library(DAAG)
m.glm <- glm(model, data=data2, family='binomial')
CVbinary(m.glm, rand=NULL, nfolds=k, print.details=TRUE)##
## Fold: 10 7 6 3 9 4 2 8 5 1
## Internal estimate of accuracy = 0.775
## Cross-validation estimate of accuracy = 0.675
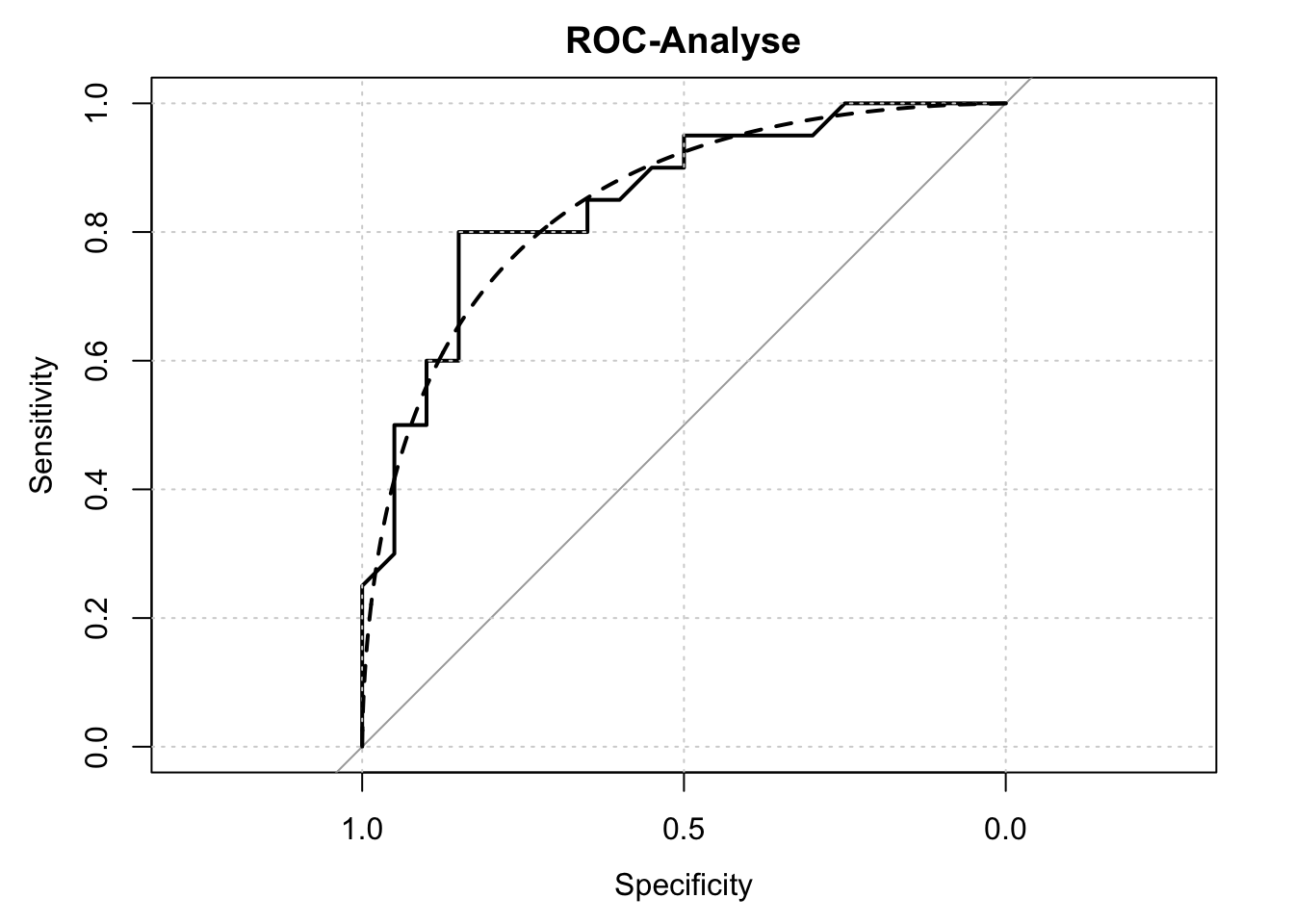
ROC-Analyse
# Prädiktor spezifizieren
# Entweder die Bezeichnung der Prädiktorvariable im Datensatz
# oder den Vektor mit den vorhergesagten Wahrscheinlichkeiten (pr) angeben
library(pROC)
predictor <- pr
response <- data2[,variablen[1]]
ROC <- roc(response, predictor, levels=levels(data2[,variablen[1]]), ci=TRUE); ROC## Setting direction: controls < cases##
## Call:
## roc.default(response = response, predictor = predictor, levels = levels(data2[, variablen[1]]), ci = TRUE)
##
## Data: predictor in 20 controls (response negativ) < 20 cases (response positiv).
## Area under the curve: 0.8488
## 95% CI: 0.7282-0.9693 (DeLong)plot(ROC, main="ROC-Analyse"); grid()
plot.roc(smooth(ROC), add=TRUE, lty=2)